
「RとStanで始めるベイズ統計モデリングによるデータ分析入門」「実践編第5部第9章 動的一般化線形モデル:ポアソン分布を仮定した例」を対象に,公開されているR,Stanのコードをpython,pystanのコードへと書き直した一例です。Stanの代わりにpystanを利用しています。
この章では,ポアソン分布を仮定したDGLMの実装例が紹介されています。
本ページでは公開されていない書籍の内容については一切触れません。理論や詳しい説明は書籍を参照してください。
なお,こちらで紹介しているコードには誤りが含まれる可能性があります。内容やコードについてお気づきの点等ございましたら,ご指摘いただけると幸いです。
(本ページにて紹介しているコードはgithubにて公開しています。)
DataAnalOji.hatena.sample/python_samples/stan/5-9-動的一般化線形モデル:ポアソン分布を仮定した例.ipynb at master · Data-Anal-Ojisan/DataAnalOji.hatena.sample
samples for my own blog. Contribute to Data-Anal-Ojisan/DataAnalOji.hatena.sample development by creating an account on GitHub.
分析の準備
パッケージの読み込み
plotSSM関数についてはこちらをご参照ください。
import arviz
import pystan
import numpy as np
import pandas as pd
%matplotlib inline
import matplotlib.pyplot as plt
plt.style.use('ggplot')
plt.rcParams['font.family'] = 'Meiryo'
import seaborn as sns
# 自作のplotSSM関数を読み込み
from plotSSM import plotSSMデータの読み込み
fish_ts = pd.read_csv('5-9-1-fish-num-ts.csv')
fish_ts['date'] = pd.to_datetime(fish_ts['date'])
fish_ts.head(n=3)

図示
plt.figure(figsize=(10,5))
plt.plot(fish_ts['fish_num'], color='black')
plt.show()

モデルの推定
データの準備
data_list = dict(y=fish_ts['fish_num'],
ex=fish_ts['temperature'],
T=len(fish_ts))モデルの推定
# stanコードの記述(5-8-1-dglm-binom.stan)
stan_code = '''
data {
int T; // データ取得期間の長さ
vector[T] ex; // 説明変数
int y[T]; // 観測値
}
parameters {
vector[T] mu; // 水準+ドリフト成分の推定値
vector[T] r; // ランダム効果
real b; // 係数の推定値
real<lower=0> s_z; // ドリフト成分の変動の大きさを表す標準偏差
real<lower=0> s_r; // ランダム効果の標準偏差
}
transformed parameters {
vector[T] lambda; // 観測値の期待値のlogをとった値
for(i in 1:T) {
lambda[i] = mu[i] + b * ex[i] + r[i];
}
}
model {
// 時点ごとに加わるランダム効果
r ~ normal(0, s_r);
// 状態方程式に従い、状態が遷移する
for(i in 3:T) {
mu[i] ~ normal(2 * mu[i-1] - mu[i-2], s_z);
}
// 観測方程式に従い、観測値が得られる
for(i in 1:T) {
y[i] ~ poisson_log(lambda[i]);
}
}
generated quantities {
// 状態推定値(EXP)
vector[T] lambda_exp;
// ランダム効果除外の状態推定値
vector[T] lambda_smooth;
// ランダム効果除外、説明変数固定の状態推定値
vector[T] lambda_smooth_fix;
lambda_exp = exp(lambda);
lambda_smooth = exp(mu + b * ex);
lambda_smooth_fix = exp(mu + b * mean(ex));
}
'''
# モデルのコンパイル
stan_model = pystan.StanModel(model_code=stan_code)
# サンプリング
dglm_poisson = stan_model.sampling(data=data_list,
seed=1,
iter=8000,
warmup=2000,
thin=6,
control={
'adapt_delta': 0.99,
'max_treedepth': 15
},
n_jobs=1)推定されたパラメタ
print(
dglm_poisson.stansummary(pars=["s_z", "s_r", "b", "lp__"],
probs=[0.025, 0.5, 0.975]))Inference for Stan model: anon_model_3e7f96660af24f75fe7e27f7ed8f7131.
4 chains, each with iter=8000; warmup=2000; thin=6;
post-warmup draws per chain=1000, total post-warmup draws=4000.
mean se_mean sd 2.5% 50% 97.5% n_eff Rhat
s_z 0.06 1.0e-3 0.03 0.02 0.05 0.14 985 1.0
s_r 0.17 4.0e-3 0.09 0.02 0.16 0.37 530 1.01
b 0.08 5.0e-4 0.02 0.04 0.08 0.12 1924 1.0
lp__ 980.59 1.39 24.71 941.01 977.14 1043.1 317 1.01
Samples were drawn using NUTS at Sun Sep 13 16:00:36 2020.
For each parameter, n_eff is a crude measure of effective sample size,
and Rhat is the potential scale reduction factor on split chains (at
convergence, Rhat=1).参考:収束の確認
# 収束確認用のRhatのプロット関数
def mcmc_rhat(dataframe, column='Rhat', figsize=(5, 10)):
plt.figure(figsize=figsize)
plt.hlines(y=dataframe[column].sort_values().index,
xmin=1,
xmax=dataframe[column].sort_values(),
color='b',
alpha=0.5)
plt.vlines(x=1.05, ymin=0, ymax=len(dataframe[column]), linestyles='--')
plt.plot(dataframe[column].sort_values().values,
dataframe[column].sort_values().index,
marker='.',
linestyle='None',
color='b',
alpha=0.5)
plt.yticks(color='None')
plt.tick_params(length=0)
plt.xlabel(column)
plt.show()
# 各推定結果のデータフレームを作成
summary = pd.DataFrame(dglm_poisson.summary()['summary'],
columns=dglm_poisson.summary()['summary_colnames'],
index=dglm_poisson.summary()['summary_rownames'])
# プロット
mcmc_rhat(summary)

print('hmc_diagnostics:\n',
pystan.diagnostics.check_hmc_diagnostics(dglm_poisson))hmc_diagnostics:
{'n_eff': True, 'Rhat': True, 'divergence': True, 'treedepth': True, 'energy': True}参考:トレースプロット
’lp__’(log posterior)のトレースプロットは図示できないため除いています。
arviz.plot_trace(dglm_poisson, var_names=["s_z", "s_r"], legend=True)

参考:推定結果一覧
print(dglm_poisson.stansummary(probs=[0.025, 0.5, 0.975]))出力が非常に多いので割愛します。
推定結果の図示
MCMCサンプルの取得
mcmc_sample = dglm_poisson.extract()個別のグラフの作成
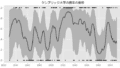
fig, ax = plt.subplots(3, 1, figsize=(15, 15))
p_all = plotSSM(mcmc_sample=mcmc_sample,
time_vec=fish_ts['date'],
obs_vec=fish_ts['fish_num'],
state_name='lambda_exp',
graph_title='状態推定値',
y_label='釣獲尾数',
axes=ax[0])
p_smooth = plotSSM(mcmc_sample=mcmc_sample,
time_vec=fish_ts['date'],
obs_vec=fish_ts['fish_num'],
state_name='lambda_smooth',
graph_title='ランダム効果を除いた状態推定値',
y_label='釣獲尾数',
axes=ax[1])
p_fix = plotSSM(mcmc_sample=mcmc_sample,
time_vec=fish_ts['date'],
obs_vec=fish_ts['fish_num'],
state_name='lambda_smooth_fix',
graph_title='気温を固定した状態推定値',
y_label='釣獲尾数',
axes=ax[2])
plt.show()

pystanの詳細については公式ページを参照してください。
PyStan — pystan 3.10.0 documentation


コメント